Bioinformatisk prosedyre på plass for analyse av bakterienes arvestoff
Forskere ved Veterinærinstituttet har etablert en bioinformatisk prosedyre for å analysere hele bakterienes arvestoff. Prosedyren kan blant annet brukes til å undersøke og identifisere antibiotikaresistens og sykdomsfremkallende egenskaper, samt å undersøke bakterienes slektskap.
Dette ble presentert på årsmøte i prosjektet QREC-MaP (Kinolonresistens til tross for lavt forbruk av antimikrobielle midler – mekanismer og mulige forebyggende tiltak). Prosjektet er finansiert av forskningsmidlene for jordbruk og matindustri (NFR 255383), og hadde nylig sitt årsmøte med representanter fra referansegruppen tilstede.
- Etablering av denne prosedyren er svært viktig for gjennomføring av prosjektet, sier prosjektleder og seniorforsker Anne Margrete Urdahl ved Veterinærinstituttet. Hun forklarer videre at mye av dette arbeidet er gjort i forbindelse med et annet forskningsprosjekt på kinolonresistente E. coli (QREC-Risk) som også er finansiert av forskningsmidlene for jordbruk og matindustri (NFR 244140).
- Denne prosedyren vil imidlertid ha synergieffekter langt utover disse to prosjektene, og bli et nyttig verktøy for Veterinærinstituttet framover. Analyse av hele bakterienes arvestoff blir mer og mer aktuelt som verktøy i mange sammenhenger, fortsetter Urdahl.
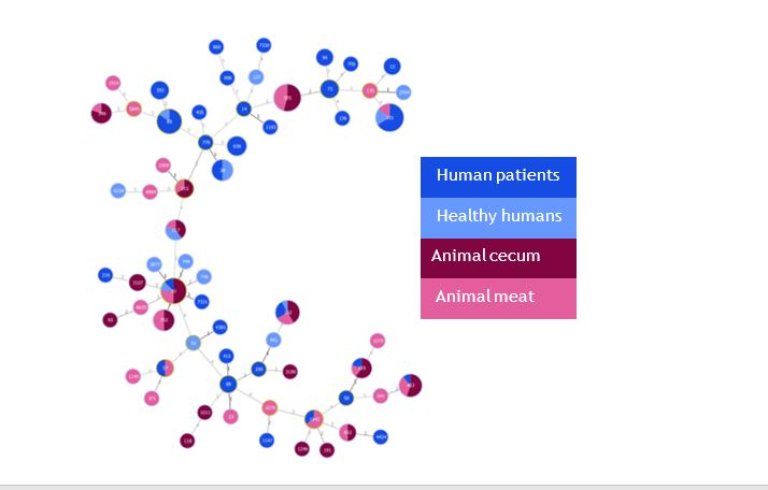
På årsmøtet ble det vist hvordan denne bioinformatiske prosedyren kan benyttes til å sammenlikne bakterier fra dyr og mat med bakterier fra syke og friske mennesker, samt for å identifisere ulike antibiotikaresistensgener.
Figuren under viser en slik sammenlikning av bakterier, hvor de blå fargene representerer bakterier fra mennesker og de rosa fra dyr/mat, og størrelsen på sirkelen gjenspeiler antall bakterier. I denne figuren er noen få av bakteriene fra mennesker og dyr/mat så nært beslektet at de kan se ut til å komme fra samme opphav.